Позиционирование нуклеосом на ДНК
Добавил пользователь Валентин П. Обновлено: 27.01.2026
Позиционирование нуклеосом — определение положения нуклеосом на последовательности ДНК эукариот. Участки ДНК могут либо входить в нуклеосомные комплексы, либо быть в составе линкерной, межнуклеосомной ДНК. Эта характеристика ДНК определяет её доступность для взаимодействия с белками.
Начало развития метода
Разработка методов по позиционированию нуклеосом берет своё начало в 1973 году, тогда было показано свойство ядерного хроматина под действием эндогенных нуклеаз расщепляться на серию отдельных фрагментов ДНК приблизительно равной длины. Первоначально в экспериментах в качестве нуклеазы использовалась дезоксирибонуклеаза I, впоследствии широкое распространение получила микрококковая нуклеаза.
Позиционирование нуклеосом при использовании микрококковой нуклеазы
Классический подход
Экспериментальный метод определения участков ДНК, входящих в состав нуклеосом, основан на применении микрококковой нуклеазы (метод MNase-seq). Клетки замораживают и измельчают, дают оттаять. После оттаивания содержащий хроматин гомогенат обрабатывают микроккоковой нуклеазой. Нуклеаза расщепляет линкерные, не защищённые нуклеосомами участки ДНК. При помощи протеаз и РНКаз раствор очищают от белков и РНК. ДНК экстрагируется из раствора. Применяется как колоночная хроматография, так варианты жидкостной экстракции — фенол-хлороформная экстракция.
ДНК осаждается этанолом. Не все линкерные участки могли расщепиться нуклеазой. На этой стадии ДНК-продукт состоит из смеси ДНК фрагментов, соответствующих участкам, с различным покрытием нуклеосомами. Фрагменты ДНК разделяются по размеру электрофорезом на агарозном геле. ДНК экстрагируется из полоски геля, содержащей мононуклеосомный продукт. Полученные фрагменты ДНК приготавливают к секвенированию. Распространено применение высокоэффективной технологии SOLiD. В результате секвенирования получают библиотеку ДНК-фрагментов. Фрагменты картируются на геном. По величине покрытия различных участков генома судят о расположении нуклеосом по последовательности ДНК.
Использование именно микроккоковой нуклеазы обусловлено тем, что она обладает как эндонуклеазной, так и экзонуклеазной активностями. Нуклеаза разрезает ДНК и последовательно отщепляет нуклеотиды со свободных концов. Недостатком микроккоковой нуклеазы является предпочтение ею, как эндонуклеазы, сайтов, составленных из чередующихся нуклеотидов dA и dT, следующими за dC или dG. В то же время она плохо распознаёт сайты из только повторяющихся dA или dT (4—6). Как экзонуклеаза микрококковая нуклеаза медленнее отщепляет нуклеотиды dC и dG. Работа фермента сильно тормозится на участках ДНК, взаимодействующих с белками. При большом времени выдержки нуклеаза начинает щепить и нуклеосомную ДНК, преимущественно в местах близости к поверхности нуклеосомы. Работа фермента в эксперименте останавливается добавлением раствора ЭДТА.
Метод MNase-seq может дополняться ультразвуковым разрушением и/или иммунопреципитацией гистона H3 (ChIP-seq), а также использованием таких ферментов, как ДНКаза (DNase-seq), транспозаза (ATAC-seq) и CpG-метилтрансфераза (NOME-seq). Может использоваться непосредственное химическое разрушение ДНК между нуклеосомами, например, при помощи гидроксильного радикала или малых ароматических молекул вроде метидиумпропил-ЭДТА, который внедряется в двойную спираль ДНК преимущественно в участках между нуклеосомами и разрушает её в присутствии катионов Fe2+ (метод MRE-seq).
Позиционирование нуклеосом in vitro
При изучении различных факторов, влияющих на распределение нуклеосом по ДНК, обычно стараются выделить компоненту влияния собственно последовательности нуклеотидов. Для этого комплекс ДНК с белками гистонами собирается in vitro из предварительно очищенной от белков ДНК и гистоновых октамеров. Количества ДНК и гистонов берутся в определённом соотношении, приблизительно один гистоновый октамер на 850 пар оснований. Далее производятся те же манипуляции, что и в обычном методе с использованием микроккоковой нуклеазы.
Предсказание сайтов связывания нуклеосом
В настоящее время развиваются методы позиционирования нуклеосом методами биоинформатики. Основа методов — экспериментально выявленные закономерности распределения нуклеосом в различных областях генома, и зависимости этих распределений от последовательности нуклеотидов ДНК. Данные по результатам экспериментального картирования нуклеосом собраны в базе данных NPRD (англ. Nucleosome Positioning Region Database).
Позиционирование нуклеосом на ДНК
Позиционирование нуклеосом на ДНК
• Нуклеосомы могут располагаться в определенных местах. Это происходит вследствие особенностей локальной структуры ДНК либо белков, взаимодействующих с определенными последовательностями
• Наиболее частая причина позиционирования нуклеосом заключается в установлении границы с участием белков, связывающихся с ДНК
• Позиционирование определяет, какие регионы ДНК находятся в линкерной области, и какой стороной она располагается на поверхности нуклеосом
Известно, что in vitro нуклеосомы организуются независимо от первичной структуры ДНК, однако это не означает, что такая же ситуация имеет место in vivo. Всегда ли in vivo определенная последовательность в ДНК находится на определенном месте относительно расположения нуклеосомы? Или же нуклеосомы расположены на ДНК случайным образом, и определенная последовательность может находиться в любом ее участке, например в коровой части в одной копии генома и в линкерной, в другой его копии?
Для ответа на этот вопрос необходимо установить положение определенной последовательности в ДНК, точнее, необходимо установить положение определенного участка ДНК по отношению к нуклеосоме. иллюстрирует принцип используемого метода.
Предположим, что последовательность ДНК организована в нуклеосомы в только одной специфической конфигурации таким образом, что каждый сайт ДНК всегда локализован на нуклеосоме в специфическом положении. Такой тип организации называется позиционированием (или иногда фазированием) нуклеосомы. В серии позиционированных нуклеосом линкерные области ДНК включают уникальные сайты.
Рассмотрим последовательность, находящуюся в пределах одной нуклеосомы. При обработке микрококковой нуклеазой высвобождаются мономерные фрагменты, в том числе фрагмент, содержащий специфическую последовательность. Если этот фрагмент обработать рестриктазой, которая имеет только один сайт узнавания, то он должен разрезаться по специфическому месту. При этом образуются два фрагмента определенной длины.
Фрагменты ДНК, полученные после переваривания микрококковой нуклеазой и рестриктазой, фракционируют методом электрофореза в геле. Для идентификации соответствующего фрагмента в «двойном переваре» используется проба, представляющая собой последовательность на одном конце сайта рестрикции. Такая техника носит название метода гибридизационной концевой метки.
Таким образом, наличие одной полосы свидетельствует о том, что найдено положение сайта рестрикции по отношению к концу нуклеосомной ДНК (по месту ее атаки микрококковой нуклеазой), и нуклеосома позиционирована на определенной последовательности ДНК
Что происходит, если нуклеосомы не занимают определенного положения? В этом случае в каждой копии генома линкеры содержат различные последовательности ДНК. Таким образом, каждый раз сайт рестрикции находится на разном месте; фактически он может занимать любое местоположение относительно концов мономерной нуклеосомной ДНК Рисунок ниже показывает, что в результате двойного разрыва в ДНК образуется набор фрагментов, варьирующих по длине от наименьшего детектируемого (20 пн) до полного фрагмента мономерной ДНК.
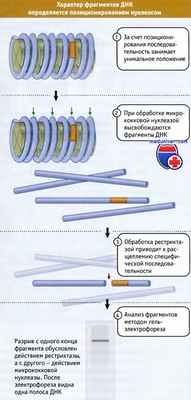
За счет позиционирования нуклеосом сайты рестрикции занимают уникальное положение по отношению к линкерным участкам,
расщепляемым микрококковой нуклеазой.
Позиционирование нуклеосом может осуществляться одним из двух способов:
• Внутренний: каждая нуклеосома специфически формируется в определенной позиции нуклеотидной последовательности. Это меняет нашу точку зрения на нуклеосому, как на субъединицу, способную образовываться при взаимодействии октамера гистонов с любой последовательностью ДНК
• Внешний: первая нуклеосома предпочтительно собирается в определенной позиции региона. Предпочтительная исходная точка для позиционирования нуклеосомы задается регионом, из которого исключаются нуклеосомы. Регион исключения обеспечивает границу, которая ограничивает позиции, доступные для соседних нуклеосом. Затем последовательно может собираться серия нуклеосом, которая характеризуется определенной длиной повтора.
В настоящее время стало очевидным, что расположение октамеров гистонов на ДНК не является случайным по отношению к ее первичной структуре. В одних случаях позиционирование носит внутреннюю природу и определяется структурными характеристиками ДНК В других случаях оно является внешним и зависит от взаимодействия других белков с ДНК и/или с гистонами.
На позиционирование октамеров гистонов влияют некоторые особенности структуры ДНК. Молекуле ДНК присуща тенденция сгибаться преимущественно в одном направлении. Так, АТ-богатые участки локализованы таким образом, что малая бороздка находится со стороны октамера гистонов, а ГЦ-богатые участки расположены так, что она оказывается снаружи.
Длинные участки дА-дТ (> 8 пн) не позиционируются в центральном суперспиральном витке вокруг коровой частицы. Пока еще не представляется возможным учесть все структурные особенности ДНК и предсказать расположение определенной ее последовательности по отношению к нуклеосоме. Также могут играть роль последовательности, которые ответственны за экстремальные процессы, например такие, как исключение нуклеосом, в результате чего могут проявляться граничные эффекты.
Обычно нуклеосомы позиционируются поблизости от границы региона. Если в их строении наблюдается некоторая вариабельность, например длина линкера изменяется, скажем, на 10 пн, то, начиная с первой определенной нуклеосомы, находящейся на границе, специфичность их дальнейшего расположения снижается. В этом случае можно ожидать, что позиционирование будет поддерживаться только для нуклеосом, расположенных относительно близко к границе.
Расположение ДНК в нуклеосоме можно описать с помощью двух моделей. На рисунке ниже показана модель трансляционного позиционирования, которое описывает положение ДНК относительно границы нуклеосомы. В особенности это относится к последовательностям, находящимся в линкерной области. Сдвиг ДНК на 10 пн вызывает образование следующего витка в линкерной области. Таким образом, трансляционное позиционирование определяет, какие регионы ДНК являются более доступными (по крайней мере по отношению к микрококковой нуклеазе).
Поскольку ДНК расположена на поверхности октамера гистонов, одна из сторон любой последовательности закрыта белком, а другая остается открытой. В зависимости от расположения на нуклеосоме, сайт ДНК, который должен узнаваться регуляторным белком, может быть доступным или недоступным. Поэтому, важную роль играет точное расположение октамера гистонов по отношению к последовательности ДНК. На рисунке ниже изображена схема ротационного позиционирования двойной спирали по отношению к поверхности коровой частицы. Если ДНК сдвигается на неполный оборот (вообразите, что ДНК вращается по отношению к белковой поверхности), то изменяется степень выхода последовательности за пределы коровой частицы.
Трансляционное и ротационное позиционирование может играть важную роль в обеспечении доступности ДНК. К числу наиболее полно охарактеризованных случаев позиционирования относится специфическое размещение нуклеосом на промоторных участках. Трансляционное позиционирование и/или исключение нуклеосом из определенной последовательности может оказаться необходимым для образования транскрипционного комплекса.
Некоторые регуляторные факторы могут связываться с ДНК лишь в участках, свободных от коровых частиц, поскольку доступность ДНК должна быть максимальной. Это создает границу для трансляционного позиционирования. В других случаях регуляторные факторы могут связываться с ДНК на поверхности нуклеосомы, однако ротационное позиционирование важно для открытия той стороны ДНК, на которой расположены соответствующие контактные точки.
При отсутствии позиционирования нуклеосом сайты рестрикции в различных копиях генома располагаются на разных местах.
При обработке ДНК рестриктазой, атакующей специфические последовательности (выделены красным),
и микрококковой нуклеазой, действующей на межнуклеосомные сайты (выделены зеленым),
образуется смесь разных по размеру фрагментов. Трансляционное позиционирование отражает линейное положение ДНК относительно октамера гистонов.
Смещение ДНК на 10 пн приводит к смещению последовательностей, находящихся в линкерных областях.
Однако это не влечет за собой изменений ориентации сторон ДНК,
т. е. та сторона ДНК, которая примыкала к гистонам, сохраняет свое положение. Ротационное позиционирование отражает положение сторон ДНК относительно поверхности нуклеосомы.
Любое перемещение, которое отличается от шага спирали (около 10,2 пн на виток),
приводит к смещению ДНК относительно поверхности гистонового кора.
Последовательности, расположенные внутри, в большей степени защищены от нуклеаз, чем расположенные снаружи.
Редактор: Искандер Милевски. Дата обновления публикации: 18.3.2021
Хроматиновые белки ADF1 и BEAF-32 влияют на позиционирование нуклеосом и упаковку ДНК междиска 61С7/С8 политенных хромосом Drosophila melanogaster
Формирование интерфазной хромосомы представляет собой многоуровневый процесс, в результате которого ДНК упаковывается в тысячи раз. На первом этапе упаковки образуются нуклеосомы – базовые повторяющиеся единицы хроматина. Дальнейшая упаковка происходит за счет связывания ДНК с гистоном Н1 и негистоновыми белками, участвующими в ближних и дальних энхансер-промоторных и инсуляторных взаимодействиях. При этом функциональность генома сохраняется за счет динамичной и неравномерной упаковки ДНК вдоль хромосомы, что проявляется уже на нуклеосомном уровне. Несмотря на долгую историю изучения процесса упаковки ДНК в интерфазном ядре, до сих пор до конца не ясно, от чего зависит степень упаковки разных участков ДНК и какое влияние оказывают друг на друга разные уровни упаковки. Превосходной модельной системой для изучения молекулярных механизмов, определяющих степень упаковки ДНК, являются политенные хромосомы слюнных желез личинок дрозофилы. За счет больших размеров и характерного диск/междискового рисунка они позволяют легко наблюдать неравномерность упаковки ДНК вдоль хромосом. В настоящей работе мы исследовали, какую роль играют негистоновые регуляторные белки ADF1 и BEAF-32 в позиционировании нуклеосом и формировании междиска 61С7/С8 – одного из декомпактных районов политенных хромосом. ADF1 – специфический транскрипционный фактор, а BEAF-32 – инсуляторный белок, ассоциированный с междисками. С использованием трансгенных линий мы показали, что мутации сайтов связывания ADF1 или BEAF-32 приводят к тому, что трансген теряет способность формировать междиск в новом генетическом окружении. Кроме того, мутации нарушают нуклеосомную организацию трансгена, характеризующуюся повышением стабильности нуклеосом. Мы обнаружили, что сайты связывания ADF1 и BEAF-32 необходимы для спасения нуль-аллеля bantam – жизненно важного гена микроРНК, расположенного в районе 61С7/С8. Таким образом, мы можем проследить связь между степенью упаковки ДНК, нуклеосомной организацией и функцией конкретного участка интерфазной хромосомы.
Ключевые слова
Об авторах
Институт молекулярной и клеточной биологии Сибирского отделения Российской академии наук
Россия
Новосибирск.
Институт молекулярной и клеточной биологии Сибирского отделения Российской академии наук.
Россия
Новосибирск.
Институт молекулярной и клеточной биологии Сибирского отделения Российской академии наук.
Россия
Новосибирск.
Список литературы
2. Berkaeva M., Demakov S., Schwartz Y.B., Zhimulev I. Functional analysis of Drosophila polytene chromosomes decompacted unit: the interband. Chromosome Res. 2009;17(6):745-754. DOI 10.1007/ s10577-009-9065-7.
3. Brennecke J., Hipfner D.R., Stark A., Russell R.B., Cohen S.M. Bantam encodes a developmentally regulated microRNA that controls cell proliferation and regulates the proapoptotic gene hid in Drosophila. Cell. 2003;113(1):25-36.
4. Chereji R.V., Clark D.J. Major determinants of nucleosome position-ing. Biophys. J. 2018;114(10):2279-2289. DOI 10.1016/j.bpj.2018. 03.015.
5. Chereji R.V., Kan T.-W., Grudniewska M.K., Romashchenko A.V., Be- rezikov E., Zhimulev I.F., Guryev V., Morozov A.V., Moshkin Y.M. Genome-wide profiling of nucleosome sensitivity and chromatin ac¬cessibility in Drosophila melanogaster. Nucleic Acids Res. 2016; 44(3):1036-1051. DOI 10.1093/nar/gkv978.
6. England B.P., Admon A., Tjian R. Cloning of Drosophila transcription factor Adf-1 reveals homology to Myb oncoproteins. Proc. Natl. Acad. Sci. USA. 1992;89(2):683-687.
7. Gilbert M.K., Tan Y.Y., Hart C.M. The Drosophila boundary element- associated factors BEAF-32A and BEAF-32B affect chromatin structure. Genetics. 2006;173(3):1365-1375. DOI 10.1534/genetics. 106.056002.
9. Kvon E.Z., Demakov S.A., Zhimulev I.F. Chromatin decompaction in the interbands of Drosophila polytene chromosomes does not cor¬relate with high transcription level. Russ. J. Genet. 2011;47(6):674- 681. DOI 10.1134/S1022795411060135. (in Russian)
10. Kwon S.Y., Grisan V, Jang B., Herbert J., Badenhorst P. Genome¬wide mapping targets of the metazoan chromatin remodeling factor NURF reveals nucleosome remodeling at enhancers, core promoters and gene insulators. PLoS Genet. 2016;12(4):1-26.
11. Martin F.A., Perez-Garijo A., Moreno E., Morata G. The brinker gra-dient controls wing growth in Drosophila. Development. 2004; 131(20):4921-4930. DOI 10.1242/dev.01385.
12. Moshkin Y.M., Chalkley G.E., Kan T.W., Reddy B.A., Ozgur Z., van Ijcken W.F.J., Dekkers D.H.W., Demmers J.A., Travers A.A., Ver- rijzer C.P. Remodelers organize cellular chromatin by counteract-ing intrinsic histone-DNA sequence preferences in a class-specific manner. Mol. Cell. Biol. 2012;32(3):675-688. DOI 10.1128/MCB. 06365-11.
13. Oh H., Irvine K.D. Cooperative regulation of growth by Yorkie and Mad through bantam. Dev. Cell. 2011;20(1):109-122. DOI 10.1016/j. devcel.2010.12.002.
14. Orsi G.A., Kasinathan S., Hughes K.T., Saminadin-Peter S., Heni- koff S., Ahmad K. High-resolution mapping defines the cooperative architecture of Polycomb response elements. Genome Res. 2014; 24(5):809-820. DOI 10.1101/gr.163642.113.
15. Peng H.W., Slattery M., Mann R.S. Transcription factor choice in the Hippo signaling pathway: homothorax and yorkie regulation of the microRNA bantam in the progenitor domain of the Drosophila eye imaginal disc. Genes Dev. 2009;23(19):2307-2319. DOI 10.1101/ gad.1820009.
16. Rath U., Ding Y., Deng H., Qi H., Bao X., Zhang W., Girton J., Johan-sen J., Johansen K.M. The chromodomain protein, Chromator, in-teracts with JIL-1 kinase and regulates the structure of Drosophila polytene chromosomes. J. Cell Sci. 2006;119(Pt 11):2332-2341.
17. Semeshin V.F., Demakov S.A., Shloma V.V., Vatolina T.Y., Gorcha-kov A.A., Zhimulev I.F. Interbands behave as decompacted autono¬mous units in Drosophila melanogaster polytene chromosomes. Ge- netica. 2008;132(3):267-279.
18. Talamillo A., Fernandez-Moreno M.A., Martinez-Azorin F., Born- stein B., Ochoa P., Garesse R. Expression of the Drosophila melano¬gaster ATP synthase a subunit gene is regulated by a transcriptional element containing GAF and Adf-1 binding sites. Eur. J. Biochem. 2004;271(20):4003-4013. DOI 10.1111/j.1432-1033.2004.04336.x.
19. Thomas G.H., Elgin S.C. Protein/DNA architecture of the DNase I hypersensitive region of the Drosophila hsp26 promoter. EMBO J. 1988;7(7):2191-2201.
20. Tsukiyama T., Becker P.B., Wu C. ATP-dependent nucleosome disrup¬tion at a heat-shock promoter mediated by binding of GAGA tran¬scription factor. Nature. 1994;367(6463):525-532. DOI 10.1038/ 367525a0.
21. Vogelmann J., Le Gall A., Dejardin S., Allemand F., Gamot A., Labesse G., Cuvier O., Negre N., Cohen-Gonsaud M., Margeat E., Nollmann M. Chromatin insulator factors involved in long-range DNA interactions and their role in the folding of the Drosophila genome. PLoS Genet. 2014;10(8):e1004544. DOI 10.1371/journal. pgen.1004544.
22. Zhimulev I.F., Semeshin V.F., Kulichkov V.A., Belyaeva E.S. Inter-calary heterochromatin in Drosophila. I. Localization and general characteristics. Chromosoma. 1982;87(2):197-228. DOI 10.1007/ BF00338489.
23. Zhimulev I.F., Zykova T.Y., Goncharov F.P., Khoroshko V.A., De- makova O.V., Semeshin V.F., Pokholkova G.V., Boldyreva L.V., Demidova D.S., Babenko V.N., Demakov S.A., Belyaeva E.S. Ge-netic organization of interphase chromosome bands and interbands in Drosophila melanogaster. PLoS One. 2014;9(7):e101631. DOI 10.1371/journal.pone.0101631
Позиционирование нуклеосом на гене неомицинфосфотрансферазы в репрессированном состоянии и при индукции экспрессии в составе дрожжевых плазмид Захарова Мария Глебовна

Захарова Мария Глебовна. Позиционирование нуклеосом на гене неомицинфосфотрансферазы в репрессированном состоянии и при индукции экспрессии в составе дрожжевых плазмид : диссертация . кандидата биологических наук : 03.00.03 / Захарова Мария Глебовна; [Место защиты: Моск. гос. ун-т им. М.В. Ломоносова].- Москва, 2008.- 159 с.: ил. РГБ ОД, 61 09-3/282
Введение к работе
В эукариотической клетке все процессы, определяемые ДНК (репликация, транскрипция, рекомбинация и т.д.), происходят на матрице, единицей которого является нуклеосома (Komberg R. D., and Lorch Y., 1999). Упаковка ДНК в нуклеосому существенно ограничивает возможности реализации ее потенциала: репрессируется узнавание нуклеотидной последовательности специфичными к последовательности белками, в том числе факторами, регулирующими транскрипцию, взаимодействие их с ДНК, ограничиваются процессы инициации и элонгации синтеза мРНК. В настоящее время стало понятным, что клетка обладает механизмами, которые позволяют преодолеть эти ограничения.
Более того, стало ясно, что сама нуклеосома является единицей модуляции
активности генов в ходе экспрессии. В отдельных нуклеосомах при этом происходит
глобальное их ремоделирование, включающее несколько десятков точечных энзиматических
модификаций гистонов или замену канонических гистонов их субтипами. Следствием этого
является как изменение структуры октамера гистонов, так и его взаимодействие с ДНК, что
приводит к изменению доступности ДНК для узнавания и взаимодействия с факторами
транскрипции. *
Свой значительный вклад в регуляцию экспрессии генов вносит феномен позиционирования нуклеосом. Позиционирование нуклеосом означает неслучайный характер распределения нуклеосом по последовательности ДНК или по отдельным участкам гена. На первом этапе изучения позиционирования нуклеосом было выявлено, что позиционирующим сигналом обладает сама последовательность ДНК. Выявлен ряд особенностей первичной структуры ДНК, благоприятствующих связыванию октамера или затрудняющих его. Показано, что по своей аффиности разные последовательности ДНК отличаются на порядки. Затем стало ясно, что нуклеосомы неслучайным образом расположены по гену, в частности, наиболее выражено позиционирование на промоторных участках гена. В настоящее время существуют несколько моделей, объясняющих как позиционирование нуклеосом на промоторах, которое блокирует узнавание ТАТА-боксов, преодолевается в ходе индукции гена. Практически не исследованы вопросы о возможных других сигналах позиционирования, в частности, наличие соседних нуклеосом, влияние соседствующих последовательностей ДНК и иных пограничных факторов.
Мы предложили тактику исследования этих проблем на дрожжевых плазмидах, в которых генно-инженерным путем осуществили смену последовательностей ДНК, соседствующих с той, на которой позиционирована нуклеосома.
Такой подход позволяет ответить на вопрос о роли самой последовательности ДНК или участка гена и возможном взаимовлиянии соседствующих нуклеосом на их позиционирование. Поскольку для исследования был выбран индуцибельный ген неомицинфосфотрансферазы, это позволило также связать позиционирование нуклеосом с функциональным состоянием гена.
Цели и задачи исследования
Целью настоящей работы было исследование позиционирования нуклеосом на индуцибелыюм гене неомицинфосфотрансферазы (NPTII) в составе дрожжевых плазмид. В процессе работы решались следующие задачи:
1. Выявить позиционирование нуклеосом на отдельных участках гена:
промоторе, структурной части, на вставке последовательности FRT и на вставке в ген
мономера сателлитной ДНК мыши.
2. Создать генно-инженерные конструкции, в которых для отдельных участков
гена менялись соседи, и оценить возможное влияние смены соседней последовательности
(и нуклеосом на них) на позиционирование нуклеосом на изучаемом участке гена.
3. Выявить возможное изменение позиционирования нуклеосом на промоторе
и других частях гена при индукции экспрессии гена галактозой.
4. Проверить возможный вклад гиперацетилирования гистонов при
ингибировании гистондеацетилаз трихостатином А в изменение позиционирования
нуклеосом.
Научная новизна работы
Предложен новый способ оценки взаимного влияния соседних последовательностей (и наличия нуклеосом на них) на позиционирование нуклеосом. Смену соседних последовательностей осуществляли генно-инженерным путем.
Показано, что нуклеосомы позиционированы на разных участках гена индивидуально и их положение не зависит от соседних последовательностей, соседних нуклеосом, и определяется позиционирующими трансляционным и пограничным сигналами.
Показано, что все нуклеосомы позиционированы поливариантно, что предполагает скольжение октамера гистонов относительно последовательности ДНК.
Установлено, что на промоторе гена NPTII в неактивном состоянии нуклеосомы закрывают ТАТА-бокс. При экспрессии гена нуклеосома смещается с ТАТА-бокса и репозиционируется. Новое положение этой нуклеосомы обеспечивает пространственное сближение энхансера и ТАТА-бокса.
Такой же эффект без воздействия активатора достигается при ингибировании деацетилаз гистонов.
Выявлена возможность двойного характера экспрессии одного и того же гена NPTII: индуцибельной с GALCYC-промотора и базальной с ТАТА-элемента FRT.
Практическая значимость работы
Результаты работы могут быть использованы в практике научных исследований в области молекулярной биологии.
Апробация работы
Основные результаты работы докладывались и обсуждались на Международном симпозиуме по проблемам мейоза (Санкт-Петербург,2003).
По материалам диссертации опубликовано 5 печатных работ.
Структура работы
Диссертация состоит из разделов «Введение», «Обзор литературы», «Материалы и методы», «Результаты и их обсуждение», «Заключение», «Выводы», «Список литературы».
научная статья по теме ПОЗИЦИОНИРОВАНИЕ НУКЛЕОСОМ НА РЕПРЕССИРОВАННОМ И АКТИВНОМ ГЕНЕ НЕОМИЦИНФОСФОТРАНСФЕРАЗЫ (NPTII) В СОСТАВЕ ДРОЖЖЕВОЙ ПЛАЗМИДЫ Биология

Текст научной статьи на тему «ПОЗИЦИОНИРОВАНИЕ НУКЛЕОСОМ НА РЕПРЕССИРОВАННОМ И АКТИВНОМ ГЕНЕ НЕОМИЦИНФОСФОТРАНСФЕРАЗЫ (NPTII) В СОСТАВЕ ДРОЖЖЕВОЙ ПЛАЗМИДЫ»
МОЛЕКУЛЯРНАЯ БИОЛОГИЯ, 2008, том 42, № 6, с. 1030-1039
МОЛЕКУЛЯРНАЯ БИОЛОГИЯ КЛЕТКИ
ПОЗИЦИОНИРОВАНИЕ НУКЛЕОСОМ НА РЕПРЕССИРОВАННОМ И АКТИВНОМ ГЕНЕ НЕОМИЦИНФОСФОТРАНСФЕРАЗЫ (NPTII) В СОСТАВЕ ДРОЖЖЕВОЙ ПЛАЗМИДЫ
© 2008 г. Г. И. Кирьянов1*, Л. В. Исаева1, Л. Н. Кинцурашвили2, М. Г. Захарова3
Научно-исследовательский институт физико-химической биологии им. АН. Белозерского Московского государственного университета им. М.В. Ломоносова, Москва, 119992 2Центр сельскохозяйственной биотехнологии Министерства образования и науки Республики Грузия,
Тбилиси, 380062 Грузия 3Сертификационный Центр "ПРОДЭКС", Москва, 119992 Поступила в редакцию и принята к печати 10.06.2008 г.
Сконструирована дрожжевая плазмида, содержащая гибридный промотор GAL-CYC, ген NPTII, кодирующий неомицинфосфотрансферазу, и расположенную между ними последовательность FRT. В GAL-CYC промоторе находятся четыре UAS-последовательности и два сближенных ТАТА-бокса в CYC-части [1]. При индукции несущих эту конструкцию клеток дрожжей галактозой ген NPTII эффективно экспрессируется, что обеспечивает устойчивость клеток к антибиотику G418. Изучали позиционирование нуклеосом на репрессированном и индуцированном гене NPTII в трансформированных клетках. В репрессированном состоянии (рост на глюкозе) отчетливо видно позиционирование трех нуклеосом. На CYC-части промотора локализованы две нуклеосомы, причем одна из них захватывает оба ТАТА-бокса; третья нуклеосома перекрывает FRT-последовательность и начало структурной части гена. Все три нуклеосомы позиционированы поливариантно, что предполагает возможность их скольжения вдоль ДНК. При активации экспрессии гена NPTII галактозой наблюдается скольжение двух нуклеосом по промотору, вследствием чего "открывается" ТАТА-бокс и экспонируется достаточно протяженный участок промотора. 5'-Дистальная нуклеосома сдвигается и оказывается вблизи UAS-последовательностей, что приводит к пространственному сближению UAS с ТАТА-боксом. Это, по-видимому, облегчает процесс сборки преинициаторного комплекса. Перемещение двух нуклеосом и их репозиционирование происходят независимо друг от друга. Вторая нуклеосома сдвигается в сторону FRT-последовательности и репозици-онируется в соответствии с ее сигналом позиционирования. Индукция экспрессии не сказывается на позиционировании нуклеосом на структурной части гена. Однонаправленное скольжение (sliding) и репозиционирование нуклеосом происходят и без участия индуктора при ингибировании деацетилаз трихоста-тином А. Показан феномен "базальной" (с меньшей эффективностью) экспрессии гена NPTII при неиндуцированном GAL-CYC-промоторе или при разобщении NPTII и GAL-CYC-промотора инверсией гена. Экспрессия гена в этих случаях, вероятнее всего, направляется с ТАТА-подобного элемента FRT-последовательности. Этот элемент локализован в области, постоянно экспонированной in vivo.
Ключевые слова: нуклеосома, позиционирование, скольжение, дрожжи, экспрессия.
NUCLEOSOME POSITIONING WITHIN NEOMYCINPHOSPHOTRANSFERASE GENE (NPTII) ON YEAST PLASMID IN REPRESSED AND ACTIVE STATE, by G. I. Kiryanov1*, L. V. Isayeva1, L. N. Kintsurashvili2, M. G. Zakharova3 (1Belozersky Institute of Physico-Chemical Biology, Moscow State University, Moscow, 119992 Russia; * e-mail: gikmsu@mail.ru; 2Center Agricultural Biotechnology, Ministry of Education and Science of Georgian Republic, Tbilisi, 380062 Georgia; Certification Center "Prodex", Moscow, 119991 Russia). Yeast recombinant plasmid containing FRT-sequence flanked by hybrid GAL-CYC promoter and NPTII gene was developed. GAL-CYC promoter contains four UAS sequences and two closely associated TATA-boxes in CYC part [1]. This construct provides galactose-inducible synthesis of neomycinphosphotransferase from NPTII gene, and, thus, resistance of
Принятые сокращения: NPTII (neomycinphosphotransferase) - неомицинфосфотрансфераза; G418 - генетицин; TSA(trichosta-tine A) - трихостатин A; UAS (upstream activating sequence) - вышележащая активаторная последовательность, энхансер; FRT (FLP recombination target) - мишень для FLP-рекомбиназы; HDAC (Histone Deacetylase) - гистондеацетилаза; HAT (His-tone Acetyltransferase) - гистон-ацетилтрансфераза.
transformed cells to G418 antibiotic. Nucleosome positioning within NPTII gene in repressed and active states was studied. Under repressive conditions (growth on glucose) stable positioning of three nucleo-somes was detected. Two nucleosomes are localized in CYC-part. One of them encompasses both TATA-boxes. The third nucleosome overlaps FRT sequence and start of NPTII gene coding sequence. All three nucleosomes show multiple positioning. It suggests possibility of nucleosome sliding along DNA. After induction of NPTII expression by galactose sliding of two nucleosomes is detected. Sliding leads to exposure of TATA-box and long promoter segment. Sliding results in stable repositioning of nucleosomes at new sites. 5'-distal nucleosome moves closer to UAS-sequences. As a results UAS becomes spatially closer to TATA-box. This proximity facilitates assembly of preinitiation complex. Nucleosomes slides independently from each other. The second nucleosome moves towards FRT-sequence and repositions at its nu-cleosome positioning signal. Galactose-induced expression does not affect nucleosome positioning with coding region of NPTII gene. Unidirectional sliding and repositioning are detected without induction after deacetylase inhibition with trichostatine A. Basal expression of NPTII gene was shown without activation of GAL-CYC promoter and after spatial uncoupling of coding sequence and promoter by gene inversion. In these cases it seems that expression is driven by TATA-like element in FRT-sequence. This element is located in permanently exposed area (in vivo data).
Key works: nucleosome, positioning, sliding, yeast, expression
В эукариотической клетке все процессы, направляемые ДНК (репликация, транскрипция, рекомбинация и т.д.), происходят на хроматиновой матрице, единицей которой является нуклеосома [2]. Упаковка ДНК в нуклеосому существенно ограничивает возможности реализации ее потенциала: репрессируется узнавание нуклеотидной последовательности специфичными к последовательности белками, в том числе факторами, регулирующими транскрипцию, взаимодействие их с ДнК, ограничиваются процессы инициации и элонгации синтеза мРНК. В настоящее время стало понятно, что клетка обладает механизмами, которые позволяют преодолеть эти ограничения.
В свете исследований, проведенных за последнее 10-летие, становится очевидным, что сама нуклеосома представляет собой единицу модуляции активности генов в ходе экспрессии. Термин "активная" нуклеосома описывает динамические изменения состояния самой нуклеосомы, октамера гистонов и отдельных участников октамера, которые определяют возможность снятия репрессивных ограничений. Открыты, широко исследуются и уточняются основные механизмы, связанные с активацией нук-леосом : АТР-зависимое ремоделирование нукле-осом, обеспечивающее изменение места посадки октамера на ДНК, обратимые ковалентные модификации гистонов октамера, изменяющие как структуру октамера и его стабильность, так и взаимодействие октамера с ДНК. В свою очередь, модификации гистонов обеспечивают изменение доступности ДНК для сайт- и последовательность-специфичных факторов.
Третий механизм изменения структуры нуклеосомы - замена гистонов их субтипами, например, гистона Н3 на Н3.3, также приводящая к образованию более релаксированной формы нуклеосомы. Роль гистонов в процессах активации нуклеосомы интерпретируется в терминах "гистонового кода".
Менее обсуждаем другой аспект регуляции активности генов, который можно рассматривать в терминах "второго кода ДНК".
Известно, что в промоторах индуцибельных генов представлены нуклеотидные последовательности, прямо определяющие сайт-специфичность взаимодействия с ними белков-активаторов. Можно привести наиболее хорошо изученные примеры: участки взаимодействия с гормон-рецепторными комплексами стероидных гормонов [3]; UAS-после-довательности (энхансеры), определяющие связывание белков-активаторов Pho2 и Pho4 (для генов, индуцируемых концентрацией фосфата), Gal4 (для генов, индуцируемых галактозой) и т.п. Следует отметить, что взаимодействие этих активаторов в клетке происходит на ДНК в составе хроматина. В ряде случаев участки узнавания активаторов находятся в составе нуклеосом и недоступны для узнавания, в других случаях белки-активаторы постоянно связаны со своими сайтами [4]. Другой аспект "второго кода ДНК" - феномен позиционирования нуклеосом. Известно, что предпочтительность связывания октамера гистонов с ДНК определяется особенностями ее структуры, в конечном итоге, нуклеотидной последовательностью [5, 6]. В настоящее время не вполне понятно, как позиционирование нуклеосом на промоторах, блокирующих узнавание ТАТА-боксов, преодолевается в ходе индукции гена. В качестве примера можно привести активацию генов PHO5, при которой происходит разборка нуклеосомы (нуклеосом), блокирующих ТАТА-бокс. В этом процессе принимают участие шапероны, удаляющие Н2А-Н2В-димеры из окта-меров [7]. Существует много примеров альтернативного решения проблемы. В опытах in vitro и in vivo показано смещение октамера гистонов с функционально значимого участка ДНК, описываемое как скольжение нуклеосом [8, 9].
Ранее мы описали позиционирование нуклеосом на модели индуцируемого галактозой промотора гена NPTII в дрожжевых плазмидах. Одно из главных наблюдений, сделанных на этих моделях, - заключение о поливариантности позиционирования нуклеосом на каждой конкретной нуклеотидной последовательности. Мы интерпретируем поливариантность как возможность скольжения нуклеосомы относительно сигнала позиционирования (аффинного или пограничного).
В представленной работе мы детализировали положение отдельных нуклеосом на GAL-CYC-ин-дуцибельном промоторе гена NPTII в репрессированном и индуцированном состоянии, оценив при этом реальный сдвиг нуклеосом. Параллельно оцен
Для дальнейшего прочтения статьи необходимо приобрести полный текст. Статьи высылаются в формате PDF на указанную при оплате почту. Время доставки составляет менее 10 минут. Стоимость одной статьи — 150 рублей.
Читайте также:
